آموزش بیوانفورماتیک؛ شبیه سازی Docking & Mol Dynamics را یاد بگیرید - آخرین آپدیت
Bioinformatics; Learn Docking & Mol Dynamics Simulation
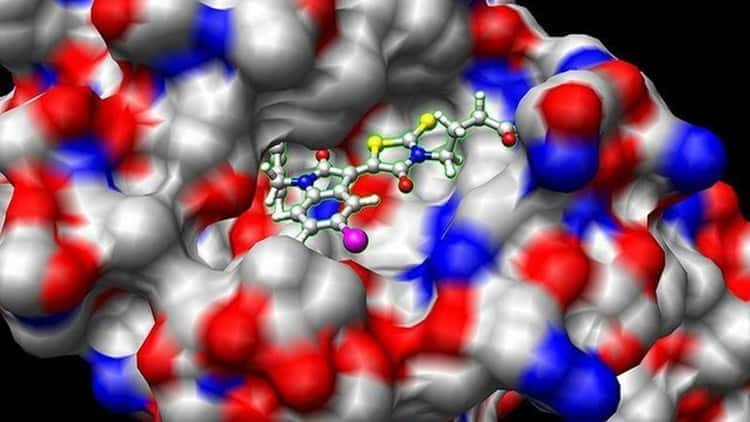
در حال حاضر، استفاده زیادی از ابزارهای بیوانفورماتیک در مطالعات بیولوژیکی وجود دارد. این دوره پیشرفته بیوانفورماتیک شامل جنبه های تئوری و عملی اتصال مولکولی و شبیه سازی دینامیک مولکولی برای شما می باشد. در این دوره سعی شده است مراحل تئوری و عملی مورد نیاز برای انجام تکنیک های بیوانفورماتیک فوق توضیح داده شود. ما قویاً معتقدیم که پس از گذراندن این دوره، شما به خوبی با اتصال لیگاند-پروتئین و شبیهسازی دینامیک مولکولی آشنا خواهید شد.
این دوره برای در نظر گرفتن نیاز زیست شناسان طراحی شده است. ما سعی کردیم هر مرحله را به روشی ساده و زیبا توضیح دهیم. مانند دوره قبلی ما، "یادگیری بیوانفورماتیک از ابتدا (تئوری عملی)" در این دوره، ما از ابتدا شروع کردیم. بنابراین، اگر در این زمینه تازه کار هستید، حرکت با جریان برای شما آسان خواهد بود. در مجموع هفت ماژول با 104 سخنرانی وجود دارد. در این دوره آموزشی
را یاد خواهید گرفت(1) بیوانفورماتیک ساختاری پروتئین
(2) نظریه شبیه سازی دینامیک مولکولی
(3) معرفی محیط لینوکس
(4) نمایش عملی شبیه سازی دینامیک مولکولی پروتئین
(5) اتصال لیگاند-پروتئین (تئوری عملی)
(6) نمایش عملی شبیه سازی دینامیک مولکولی مجتمع لیگاند-پروتئین
(7) غربالگری مجازی لیگاندها با پروتئین با استفاده از وینا
این دوره ترکیبی منحصر به فرد از تئوری و عملی است. به شما این اطمینان را می دهیم که پس از گذراندن این دوره، انجام داکینگ مولکولی و دینامیک مولکولی برای شما آسان خواهد بود. ما در این دوره از نرم افزار متن باز استفاده کرده ایم تا نیازی به خرید نرم افزار گران قیمت نداشته باشید. ابزارهایی که در این دوره نشان داده شده اند عبارتند از Modeller، I-TASSER، Alpha-Fold (Colab)، Auto-dock4، Vina، Patch-Dock، GROMACS، LigPlot، ChemSketch، OpenBabel، Pymol و بسیاری دیگر.
پس منتظر چه چیزی هستید؟ روی دکمه ثبت نام کلیک کنید و این سفر شگفت انگیز را شروع کنید. امیدواریم مطالب این دوره ارزش پول شما را داشته باشد. ما از هر کمک ممکن در طول فرآیند یادگیری شما اطمینان می دهیم. ما همیشه به سوالات و سوالاتی که شما برای ما ارسال می کنید پاسخ می دهیم. ما معتقدیم که با شما رشد خواهیم کرد.
رشد شما رشد ماست.
ماساژ مهم قبل از شروع دوره Important Massage Before Start of The Course
-
ماساژ مهم مربیان Important Massage from Instructors
(اختیاری) ماژول-1 مبانی ساختار پروتئین و روش دریافت آنها (Optional) Module-1 Protein Structure Basics & Way to Get Them
-
پروتئین چیست؟ What is Protein?
-
تکنیک های تجربی برای تعیین ساختار سه بعدی پروتئین Experimental Techniques to Determine 3D Structure of Protein
-
عملی-1: نحوه دانلود ساختار پروتئین از بانک اطلاعات پروتئین Practical-1: How To Download Protein Structure From Protein Databank
-
عملی-2: تجسم ساختار سه بعدی پروتئین با استفاده از Pymol Practical-2: Visualization of 3D Structure of Protein Using Pymol
-
رفع باقی مانده های از دست رفته در ساختار سه بعدی پروتئین دانلود شده است Fixing the Missing Residues in Protein 3D Structure Downloaded
-
عملی-3: رفع مشکل باقیمانده های گمشده با Modeller Practical-3: Fixing Missing Residues Issue With Modeller
-
مقدمه ای بر پیش بینی ساختار سه بعدی پروتئین Intro to Protein 3D Structure Prediction
-
عملی-4: نحوه پیش بینی ساختار سه بعدی با استفاده از مدلر (هومولوژی) Practical-4: How to Predict 3D Structure Using Modeller (Homology)
-
عملی-5: چگونه از I-TASSER برای پیش بینی ساختار سه بعدی استفاده کنیم؟ Practical-5: How to Use I-TASSER for Prediction of 3D Structure?
-
عملی-6: معرفی پایگاه داده آلفا فولد و کولب آلفا فولد Practical-6: Introduction of Alpha-Fold Database and Alpha-Fold Colab
-
آزمون 1: بررسی دانش ماژول-1 Quiz 1: Knowledge Check of Module-1
-
تکلیف 1: رفع باقی مانده اسیدهای آمینه از دست رفته در ساختار پروتئین Assignment-1: Fix the Missing Amino Acids Residues in the Protein Structure
ماژول-2: نظریه شبیه سازی دینامیک مولکولی Module-2: Theory of Molecular Dynamics Simulations
-
معرفی پایه شبیه سازی دینامیک مولکولی و مراحل آن Basic Introduction of Molecular Dynamic Simulations and its Steps
-
نظریه پایه شبیه سازی دینامیک مولکولی (قسمت 1) Basic Theory of Molecular Dynamics Simulation (Part-1)
-
نظریه پایه شبیه سازی دینامیک مولکولی (قسمت 2) Basic Theory of Molecular Dynamics Simulation (Part-2)
-
نظریه پایه شبیه سازی دینامیک مولکولی (بخش 3) Basic Theory of Molecular Dynamics Simulation (Part-3)
-
نظریه پایه شبیه سازی دینامیک مولکولی (قسمت 4) Basic Theory of Molecular Dynamics Simulation (Part-4)
-
چرخه MD (شبیه سازی دینامیک مولکولی). MD (Molecular Dynamics Simulation) Cycle
-
مراحل اولیه شبیه سازی دینامیک مولکولی Basic Steps of Molecular Dynamics Simulations
-
آماده سازی ساختار پروتئین برای شبیه سازی دینامیک مولکولی Protein Structure Preparation for Molecular Dynamics Simulations
-
تهیه فایل توپولوژی شبیه سازی دینامیک مولکولی Preparation of Topology File for Molecular Dynamics Simulations
-
حل و یونیزاسیون سیستم Solvation and Ionization of System
-
کمینه سازی انرژی (EM) سیستم Energy Minimization (EM) of System
-
فاز تعادل و تولید شبیه سازی دینامیک مولکولی Equilibration and Production Phase of Molecular Dynamics Simulation
-
آزمون 2: بررسی دانش ماژول 2 Quiz 2: Knowledge Check of Module 2
ماژول 3: سیستم عامل کامپیوتر مورد استفاده برای شبیه سازی دینامیک مولکولی Module 3: Computer Operating System Used for Molecular Dynamics Simulation
-
نوع پایه سیستم عامل موجود در بازار Basic Type of Operating System Available in Market
-
ویژگی های سیستم عامل لینوکس Features of Linux Operating System
-
ترمینال سیستم عامل لینوکس Terminal of Linux Operating System
-
برخی از دستورات اولیه برای سیستم عامل لینوکس Some Basic Commands for Linux Operating System
-
بعد چه می شود؟ What's Next?
-
آزمون 3: بررسی دانش ماژول-3 Quiz 3: Knowledge Check of Module-3
ماژول 4: نمایش شبیه سازی MD پروتئین در آب با GROMACS Module 4: Demonstration of MD Simulation of Protein in Water with GROMACS
-
عملی-7 نصب لینوکس در محیط ویندوز Practical-7 Installation of Linux in Windows Environment
-
عملی-8 نصب GROMACS Practical-8 Installation of GROMACS
-
ماژول های GROMACS مورد استفاده در آموزش دینامیک مولکولی Modules of GROMACS Used in Molecular Dynamics Tutorial
-
جریان کار دینامیک مولکولی در GROMACS Work Flow of Molecular Dynamics in GROMACS
-
عملی-9 ایجاد پوشه در اوبونتو برای شبیه سازی دینامیک مولکولی Practical-9 Creating Folder in Ubuntu For Molecular Dynamics Simulations
-
دستورات برای اجرای شبیه سازی دینامیک مولکولی (برای شما) Commands to Run Molecular Dynamics Simulations (For You)
-
عملی-10 پاکسازی ساختار پروتئین برای شبیه سازی دینامیک مولکولی Practical-10 Cleaning Protein Structure For Molecular Dynamics Simulations
-
عملی-11 ایجاد فایل توپولوژی در GROMACS Practical-11 Creation of Topology File In GROMACS
-
عملی-12 حل مرحله در شبیه سازی دینامیک مولکولی Practical-12 Solvation of Step in Molecular Dynamics Simulation
-
عملی-13 مرحله یونیزاسیون در شبیه سازی دینامیک مولکولی Practical-13 Ionization Step in Molecular Dynamics Simulation
-
عملی-14 کمینه سازی انرژی (EM) در GROMACS Practical-14 Energy Minimization (EM) in GROMACS
-
عملی-15 تعادل فاز اول (قسمت الف) Practical-15 Equilibration Phase-I (Part-A)
-
عملی-16 تعادل فاز اول (قسمت B) Practical-16 Equilibration Phase-I (Part-B)
-
عملی-17 تعادل فاز دوم (قسمت الف) Practical-17 Equilibration Phase-II (Part-A)
-
مرحله دوم تعادل عملی-18 (قسمت B) Practical-18 Equilibration Phase-II (Part-B)
-
آشنایی با فایل md.mdp قبل از مرحله نهایی Understanding md.mdp File Before Final Phase
-
عملی-19 در حال اجرا شبیه سازی دینامیک مولکولی فاز نهایی Practical-19 Running Molecular Dynamics Simulation Final Phase
-
تئوری پایه ای شبیه سازی دینامیک مولکولی تجزیه و تحلیل داده ها Basic Theory of Molecular Dynamics Simulation Data Analysis
-
دستورات برای تجزیه و تحلیل داده های شبیه سازی دینامیک مولکولی Commands for Analysis of Molecular Dynamics Simulation Data
-
آوردن پروتئین به مرکز قبل از تجزیه و تحلیل داده ها Bringing the Protein in Center Before Data Analysis
-
عملی-20 محاسبه RMSD Practical-20 Calculation of RMSD
-
عملی-21 محاسبه RMSF Practical-21 Calculation of RMSF
-
عملی-22 محاسبه شعاع چرخش (Rg) Practical-22 Calculation of Radius of Gyration (Rg)
-
عملی-23 محاسبه تعداد کل پیوندهای هیدروژنی (Hb) Practical-23 Calculation of Total Number of Hydrogen bonds (Hb)
-
عملی-24 محاسبه مساحت سطح قابل دسترس حلال (SASA) Practical-24 Calculation of Solvent Accessible Surface Area (SASA)
-
عملی-25 باز کردن فایل های XVG در اکسل Practical-25 Opening of XVG Files in Excel
-
عملی-25.1: تجسم مسیر MD در Pymol Practical-25.1: Visualization of MD Trajectory in Pymol
-
آزمون 4: بررسی دانش ماژول 4 Quiz 4: Knowledge Check of Module 4
ماژول 5: مبانی اتصال مولکولی (تئوری و عملی) Module-5: Fundamentals of Molecular Docking (Theory & Practical)
-
معرفی Docking-Part-1 Introduction of Docking-Part-1
-
معرفی Docking-Part-2 Introduction of Docking-Part-2
-
الگوریتم های نمونه-بخش-1 Sampling Algorithms-Part-1
-
الگوریتم های نمونه-بخش-2 Sampling Algorithms-Part-2
-
الگوریتم های نمونه گیری - قسمت 3 Sampling Algorithms-Part-3
-
الگوریتم های نمونه گیری-قسمت 4 Sampling Algorithms-Part-4
-
الگوریتم های امتیازدهی Scoring Algorithms
-
انواع داکینگ Types of Docking
-
مراحل اولیه در پروتکل Docking Basic Steps in Docking Protocol
-
تئوری مراحل اتصال-قسمت 1 Theory of Docking Steps-Part-1
-
تئوری مراحل اتصال-قسمت 2 Theory of Docking Steps-Part-2
-
عملی-26 نصب MGL Tools، Vina، AutoDock4 و Autogrid Practical-26 Installation of MGL Tools, Vina, AutoDock4 & Autogrid
-
عملی-27: نصب ابزار ترسیم لیگاند Practical-27: Installation of Ligand Drawing Tools
-
عملی-28 دانلود و آماده سازی ساختار 3 بعدی پروتئین برای داکینگ Practical-28 Downloading and Preparation of Protein 3D Structure for Docking
-
مسئله آماده سازی ساختار پروتئین در ابزار MGL Issue With Protein Structure Preparation in MGL Tools
-
عملی-29 دانلود ساختار 3 بعدی لیگاند از پایگاه داده ZINC Practical-29 Downloading of Ligand 3D Structure from ZINC Database
-
عملی-30 ترسیم ساختار لیگاند در ChemSketch Practical-30 Drawing of Ligand Structure in ChemSketch
-
عملی-31 آماده سازی مولکول لیگاند برای اتصال Practical-31 Preparation of Ligand Molecule for Docking
-
عملی-32 پیش بینی سایت فعال پروتئین Practical-32 Prediction of Active Site of Protein
-
شبکه تنظیم عملی-33 Practical-33 Setting Grid
-
گام بسیار مهم؛ مواظب اینجا باشید Very Important Step; Be Careful Here
-
عملی-34 (قسمت الف) اجرای داکینگ با وینا Practical-34 (Part-A) Performing Docking with Vina
-
عملی-34 (قسمت ب) تجسم داده های اتصال وینا Practical-34 (Part-B) Visualization of Vina Docking Data
-
Practical-35 (Part-A) انجام Docking با استفاده از AutoDock4 Practical-35 (Part-A) Performing Docking Using AutoDock4
-
عملی-35 (قسمت B) تجسم نتایج Docking AutoDock4 Practical-35 (Part-B) Visualization of AutoDock4 Docking Results
-
Practical-33 Preforming Docking with CB Dock Practical-33 Preforming Docking with CB Dock
-
عملی-34 تجزیه و تحلیل داده های اتصال Practical-34 Analyzing the Docking Data
-
عملی-35: PatchDock; برنامه اتصال مکمل شکل Practical-35: PatchDock; Shape Complementary Docking Program
-
آزمون 5: بررسی دانش ماژول 5 Quiz 5: Knowledge Check of Module 5
ماژول-6: شبیهسازی دینامیک مولکولی مجتمع متصل به پروتئین-لیگاند Module-6: Molecular Dynamics Simulations of Protein-Ligand Docked Complex
-
بزرگترین اشکال پروتکل اتصال معمولی Biggest Drawback of Conventional Docking Protocol
-
تفاوت بین شبیه سازی MD ساده در مقابل شبیه سازی پروتئین-لیگاند MD Difference Between Simple MD Simulation Vs Protein-Ligand MD Simulations
-
مراحل اولیه شبیه سازی پروتئین لیگاند MD Basic Steps of Protein-Ligand MD Simulations
-
عملی-36: قدم اول. تهیه پروتئین و لیگاند Practical-36: Very First Step; Preparation of Protein and Ligand
-
عملی-37: فایل های لازم برای شبیه سازی دینامیک مولکولی Practical-37: Necessary Files Needed For Molecular Dynamics Simulations
-
دستورات برای اجرای شبیه سازی دینامیک مولکولی لیگاند-پروتئین Commands to Run Ligand-Protein Molecular Dynamics Simulations
-
عملی-38: تهیه فایل توپولوژی پروتئین Practical-38: Preparation of Protein Topology File
-
عملی-39: تهیه فایل توپولوژی لیگاند Practical-39: Preparation of Ligand Topology File
-
عملی-40: ساخت مجتمع Practical-40: Making Complex
-
عملی-41: حل، یونیزاسیون و کمینه سازی انرژی مجتمع Practical-41: Solvation, Ionization & Energy Minimization of Complex
-
عملی-42: مهار لیگاند Practical-42: Ligand Restrain
-
عملی-43: ترموستات Practical-43: Thermostats
-
عملی-44: تعادل Practical-44: Equilibration
-
عملی-44: فاز تولید Practical-44: Production Phase
-
عملی-44: تجزیه و تحلیل Practical-44: Analysis
ماژول-7: غربالگری مجازی مواد مخدر Module-7: Virtual Screening of Drugs
-
اهمیت غربالگری مجازی Importance of Virtual Screening
-
مقدمه ای بر غربالگری مجازی Introduction to Virtual Screening
-
مراحل اساسی غربالگری مجازی Fundamental Steps of Virtual Screening
-
دستورات مورد نیاز برای اجرای غربالگری مجازی Commands Needed to Run Virtual Screening
-
عملی-46: نصب Open Babel و Auto-Dock Vina در لینوکس (اوبونتو) Practical-46: Installation of Open Babel and Auto-Dock Vina In Linux (Ubuntu)
-
عملی-47: تهیه گیرنده پروتئین برای غربالگری مجازی Practical-47: Preparation of Protein Receptor for Virtual Screening
-
عملی-48: دانلود هزاران لیگاند در یک کلیک Practical-48: Downloading of Thousands of Ligands in One Click
-
عملی-49: آماده سازی لیگاندها برای اتصال Practical-49: Preparation of Ligands for Docking
-
عملی-50: تنظیم گرید و نوشتن فایل پیکربندی Practical-50: Setting Grid and Writing of Configuration File
-
عملی-51: اتصال لیگاندها با گیرنده ها در وینا Practical-51: Docking of Ligands with Receptors in Vina
-
عملی-52: تجزیه و تحلیل Practical-52: Analysis
به روز رسانی های اخیر در زیست شناسی ساختاری برای MD و Docking Recent Updates in Structural Biology For MD and Docking
-
به روز رسانی بانک اطلاعات پروتئین Protein Databank Update
-
چگونه در Pymol تصاویر با کیفیت انتشار خوب بسازیم؟ How to make Good Publication Quality Images in Pymol?
-
تجزیه و تحلیل داده های اتصال با استفاده از LigPlot + Analysis of Docking Data Using LigPlot+
https://donyad.com/d/6897a5
 Muhammad Dujana
Muhammad Dujana
دکتر محمد دوجانا








نمایش نظرات